Extraction, annotation et évaluation de BIOMEDICA : un aperçu complet.
Un framework révolutionnaire pour l’IA biomédicale
Le développement des Modèles Langage-Vision (VLM) dans le domaine biomédical est confronté à des défis en raison du manque de datasets multimodaux, annotés et accessibles publiquement à grande échelle dans des domaines diversifiés. Alors que des datasets ont été construits à partir de la littérature biomédicale, tels que PubMed, ils se concentrent souvent étroitement sur des domaines comme la radiologie et la pathologie, négligeant des domaines complémentaires tels que la biologie moléculaire et la pharmacogénomique qui sont essentiels pour une compréhension clinique holistique. Les préoccupations en matière de confidentialité, la complexité de l’annotation de niveau expert et les contraintes logistiques entravent également la création de datasets complets. Les approches précédentes, comme ROCO, MEDICAT et PMC-15M, ont utilisé des filtrages spécifiques au domaine et des modèles supervisés pour extraire des millions de paires image-légende. Cependant, ces stratégies échouent souvent à capturer la diversité plus large des connaissances biomédicales requises pour faire avancer les VLM biomédicaux généralistes.
En plus des limitations des datasets, la formation et l’évaluation des VLM biomédicaux présentent des défis uniques. Les approches d’apprentissage contrastif, comme PMC-CLIP et BiomedCLIP, ont montré des promesses en exploitant des datasets basés sur la littérature et des modèles de transformateurs visuels pour l’alignement texte-image. Cependant, leurs performances sont limitées par des datasets plus petits et des ressources computationnelles limitées par rapport aux VLM généraux. De plus, les protocoles d’évaluation actuels, axés principalement sur les tâches de radiologie et de pathologie, manquent de standardisation et d’applicabilité plus large. La dépendance à d’autres paramètres apprenants et aux datasets étroits compromet la fiabilité de ces évaluations, soulignant le besoin de datasets évolutifs et de cadres d’évaluation robustes pour répondre aux demandes diverses des applications biomédicales avancées en vision et langage.
Une avancée majeure pour l’extraction et l’annotation de données biomédicales
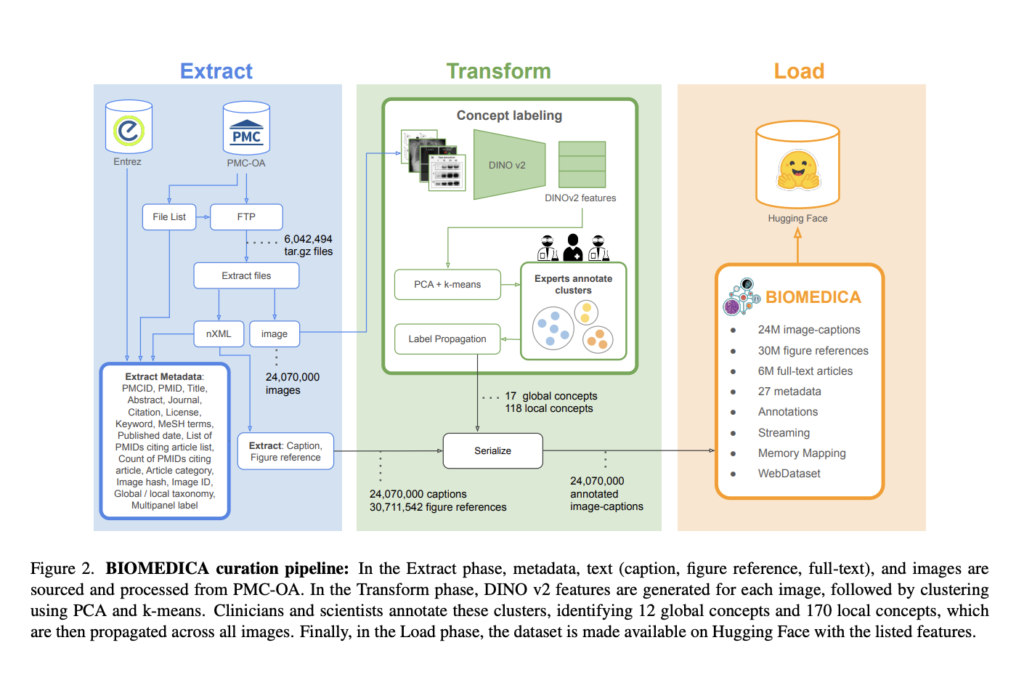
Des chercheurs de l’Université de Stanford ont introduit BIOMEDICA, un framework open-source conçu pour extraire, annoter et organiser l’ensemble PubMed Central Open Access en un dataset convivial. Cette archive comprend plus de 24 millions de paires image-texte provenant de 6 millions d’articles enrichis de métadonnées et d’annotations expertes. Ils ont également publié BMCA-CLIP, une suite de modèles de style CLIP pré-entraînés sur BIOMEDICA via streaming, éliminant le besoin de stockage local de 27 To de données. Ces modèles atteignent des performances de pointe sur 40 tâches, y compris la radiologie, la dermatologie et la biologie moléculaire, avec une amélioration moyenne de 6,56 % dans la classification zéro-shot et des besoins computationnels réduits.
Le processus de curation des données de BIOMEDICA implique l’extraction du dataset, l’étiquetage des concepts et la sérialisation. Les articles et les fichiers multimédias sont téléchargés depuis le serveur NCBI, en extrayant les métadonnées, les légendes et les références d’images à partir des fichiers nXML et de l’API Entrez. Les images sont regroupées en utilisant des embeddings DINOv2 et étiquetées à travers une taxonomie hiérarchique affinée par des experts. Les étiquettes sont attribuées par vote majoritaire et propagées à travers les clusters. Le dataset, contenant plus de 24 millions de paires image-légende et des métadonnées étendues, est sérialisé au format WebDataset pour un streaming efficace. Avec 12 concepts d’images globaux et 170 locaux, la taxonomie couvre des catégories comme l’imagerie clinique, la microscopie et les visualisations de données, mettant l’accent sur la scalabilité et l’accessibilité.
L’évaluation de l’entraînement continu sur le dataset BIOMEDICA a utilisé 39 tâches de classification biomédicale établies et un nouveau dataset de récupération de Flickr, couvrant 40 datasets. Le benchmark de classification inclut des tâches de pathologie, radiologie, biologie, chirurgie, dermatologie et ophtalmologie. Des métriques comme la précision moyenne pour la classification et le rappel de récupération (à 1, 10 et 100) ont été utilisées. Le filtrage de concepts, qui exclut les sujets sur-représentés, a mieux performé que l’équilibrage des concepts ou l’entraînement complet du dataset. Les modèles entraînés sur BIOMEDICA ont atteint des résultats de pointe, surpassant significativement les méthodes précédentes, avec des performances améliorées dans la classification, la récupération et les tâches de microscopie en utilisant moins de données et de calcul.
Une solution évolutive pour des applications biomédicales performantes
En conclusion, BIOMEDICA est un framework complet qui transforme le sous-ensemble PMC-OA de PubMed Central Open Access en le plus grand dataset prêt pour le deep learning, comportant 24 millions de paires image-légende enrichies de 27 champs de métadonnées. Conçu pour pallier le manque de datasets biomédicaux diversifiés et annotés, BIOMEDICA offre une solution évolutive et open-source d’extraction et d’annotation de données multimodales issues de plus de 6 millions d’articles. Grâce à l’entraînement continu des modèles de style CLIP utilisant BIOMEDICA, le framework atteint des performances de pointe en classification zéro-shot et en récupération image-texte sur 40 tâches biomédicales, nécessitant 10 fois moins de calculs et 2,5 fois moins de données. Toutes les ressources, y compris les modèles, les datasets et le code, sont disponibles publiquement.
Source : www.marktechpost.com







